编辑推荐:
课题组在对冷冻电镜图片进行重构分析后,观察到的主要是WTAP和VIRMA这两个辅助蛋白(A中充填部分所示),而几乎看不到METTL3和METTL14核心催化酶。
RNA的m6A甲基化修饰为RNA的所有修饰中丰度最高,广泛存在于动物、植物和微生物中,参与调控生物发育、疾病、生理等各种生物学过程。m6A的“写入”蛋白复合体由METTL3、METTL14、 WTAP、VIRMA等组成,对RNA序列(RRA*CH)进行甲基化共价修饰。对于这一复合体的结构解析,有助于解释甲基化修饰的序列选择性、位点特异性,以及“读写”的时空动态性。整个的m6A 甲基转移复合体,组成复杂、结构高度动态,单通过冷冻电镜或晶体衍射,难以目睹其全貌。正由于此,北京大学化学与分子工程学院教授、北京大学定量生物学中心PI、北大-清华生命科学联合中心PI唐淳教授课题组的合作者,华中农业大学殷平教授课题组,在对冷冻电镜图片进行重构分析后,观察到的主要是WTAP和VIRMA这两个辅助蛋白(A中充填部分所示),而几乎看不到METTL3和METTL14核心催化酶。
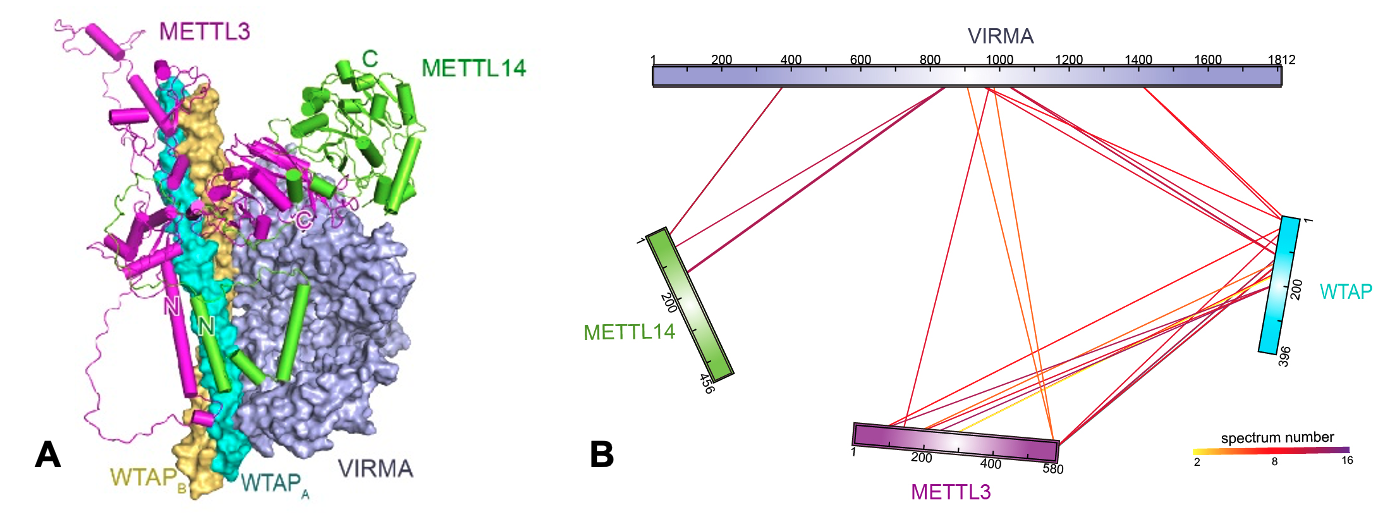
(A) RNA m6A甲基转移复合体结构;(B) 实验得到的四个蛋白之间的质谱交联关系图
唐淳一直致力于发展整合结构生物学方法。对蛋白复合体进行化学交联,然后着用高分辨质谱,就能得到相邻氨基酸之间的距离信息(B图所示)。有了这样的距离约束,就可以通过对接,获得m6A甲基转移酶中各个亚基及结构单元的大致空间关系。另一方面,利用AlphaFold2及Colabfold,可得到复合体中蛋白间的各种可能互作方式,将实验与AI预测互相比对、迭代和优化,最后就得到了RNAm6A甲基转移复合体的结构模型。这一模型可以对冷冻电镜中的电子密度给出更好的归属和解释,而核心结构酶(A中卡通所示)的催化中心紧挨着VIRMA的一侧,为后者介导的甲基化修饰的特异性提供了一种解释。这里确立的建模计算方法,基于有限的实验信息,辅以人工智能预测,也将在更多的蛋白质体系中得到应用展示。
该论文华中农业大学生科院博士研究生闫旭辉、官泽源博士和北京大学化学与分子工程学院科研助理裴凯为该论文共同第一作者,华中农业大学殷平和北京大学唐淳为共通讯作者。该工作获得了国家自然科学基金重大研究计划和科技部重点研发项目的支持。
文章链接:https://www.nature.com/articles/s41422-022-00741-8
PI简历:唐淳,北京大学化学与分子工程学院教授、北大-清华生命科学联合中心PI、北京大学定量生物学中心PI。实验室主页:http://www.tanglab.cn、http://tanglab.pku.edu.cn。
研究领域:唐淳教授课题组致力于发展基于实验数据的生物大分子结构的整合计算方法,包括生物磁共振方法和生物物理化学方法,更好的表征处于平衡态或非平衡态的生物大分子结构动态系综,分析复杂细胞环境和化学修饰对大分子结构的调控,以及动态结构变化失衡和疾病发生的关系。
生物通 版权所有