编辑推荐:
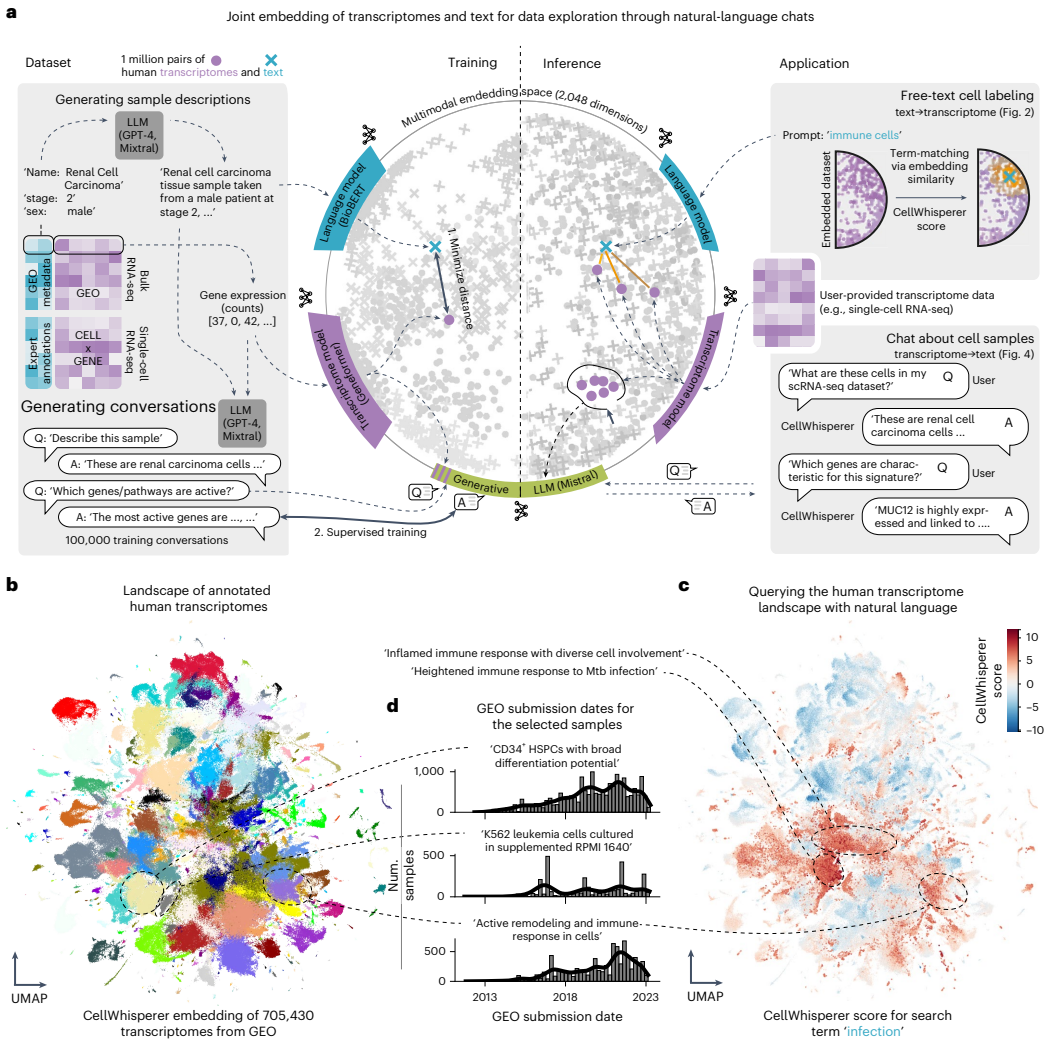
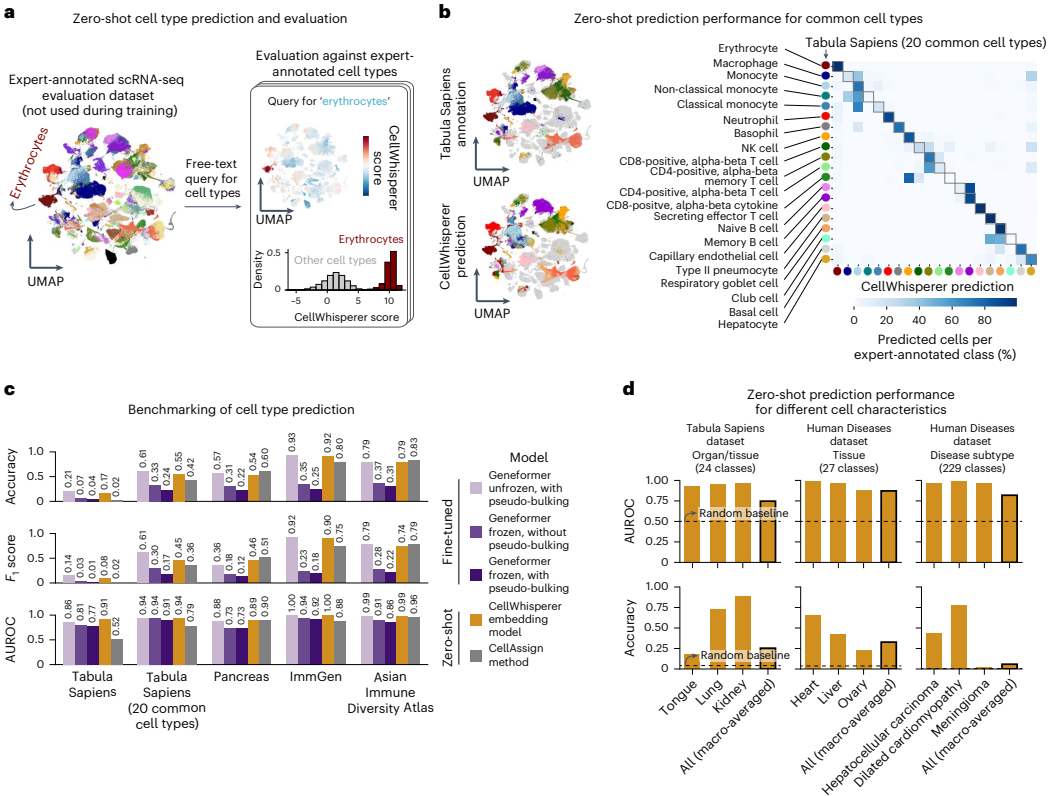
本研究针对单细胞RNA测序(scRNA-seq)数据解读复杂、依赖专业知识的挑战,开发了名为CellWhisperer的多模态人工智能模型。该模型通过对比学习整合超百万个转录组与AI生成的文本注释,构建了转录组与自然语言的联合嵌入空间,并基于微调的大语言模型(LLM)实现对话式单细胞数据交互分析。研究证实CellWhisperer在零样本预测细胞类型、疾病和组织来源等任务中表现优异,其与CELLxGENE浏览器的集成进一步降低了单细胞数据分析门槛,为生物医学研究提供了直观的AI驱动探索范式。
 生物通微信公众号
生物通微信公众号
生物通 版权所有