编辑推荐:
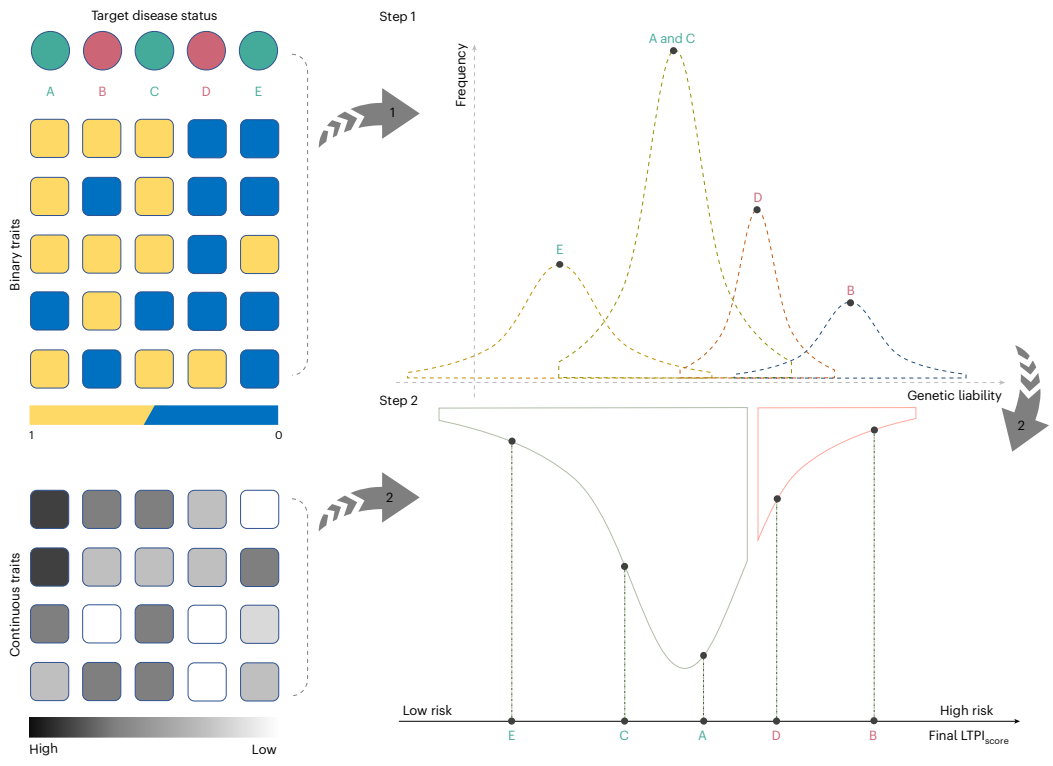
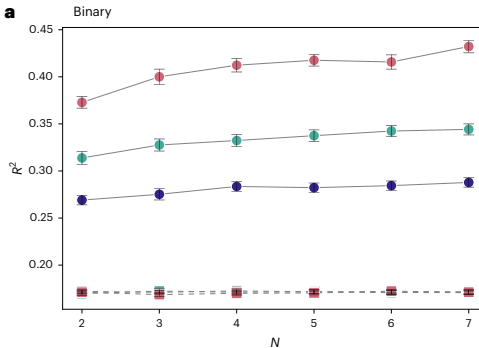
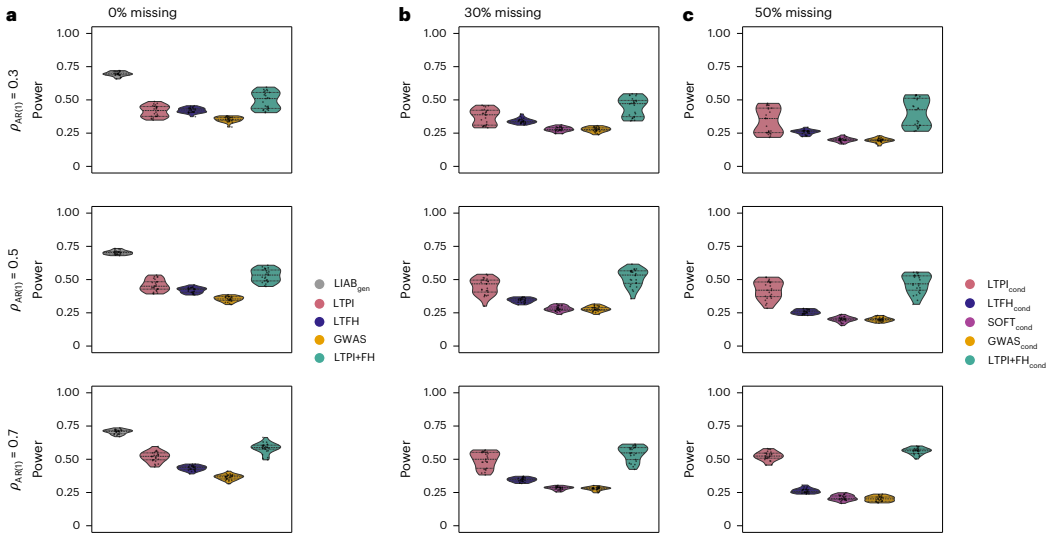
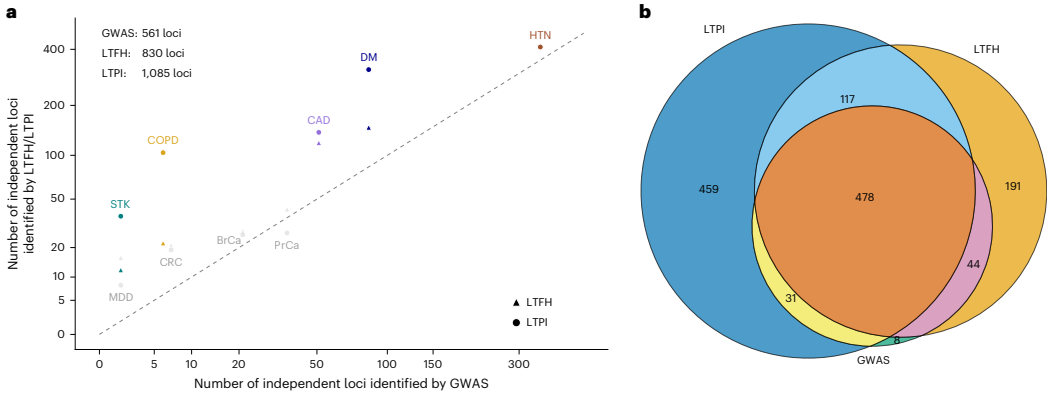
本研究针对电子健康记录(EHR)中病例-对照标签定义不精确、统计效能有限的问题,提出了责任阈值表型整合(LTPI)方法。该方法通过整合诊断代码、家族史、实验室测量值等二元/连续表型,结合遗传相关性估计,构建连续疾病风险评分。在eMERGE和UK生物样本库的应用表明,LTPI较传统PheCodes、LTFH和SoftImpute方法显著提升疾病风险预测准确性(R2达0.674)和GWAS检测功效(位点发现数增加172%),为EHR数据的精准医学应用提供新范式。
 生物通微信公众号
生物通微信公众号
生物通 版权所有