编辑推荐:
本研究针对急性白血病传统诊断方法耗时长、无法全面捕捉疾病异质性等问题,开发了基于全基因组DNA甲基化谱的快速分类框架MARLIN。研究人员构建了包含2,540例样本的参考队列,定义了38个甲基化类别,并通过神经网络实现对纳米孔测序数据的实时分析。结果显示,该方法在2小时内即可完成高精度分类(26例中25例吻合),显著优于常规诊断所需数天至数周时间,为急性白血病的精准诊疗提供了突破性技术支撑。
急性白血病是一种侵袭性血液癌症,根据造血细胞谱系可分为急性髓系白血病(AML)、B细胞淋巴母细胞白血病/淋巴瘤(B-ALL)、T细胞淋巴母细胞白血病/淋巴瘤(T-ALL)等类型。当前诊断依赖形态学、流式细胞术、核型分析和靶向测序等多重检测,通常需要数天至数周才能完成,且可能遗漏部分疾病亚型的识别。这导致治疗决策延迟,特别是对于罕见或遗传学特征不明确的病例,传统方法存在明显局限。
近年来,DNA甲基化分析在肿瘤分类中展现出巨大潜力。在中枢神经系统肿瘤领域,基于甲基化谱的分类方法不仅提高了诊断准确性,还发现了新的肿瘤类别。纳米孔测序技术的出现更进一步,能够直接检测DNA甲基化状态,无需亚硫酸氢盐转化等预处理步骤,为实时表观遗传分析提供了可能。然而,将甲基化分类应用于急性白血病仍面临两大挑战:一是需要构建涵盖所有疾病亚型的全面参考队列,二是需开发能够处理纳米孔测序初期极低覆盖度数据的计算方法。
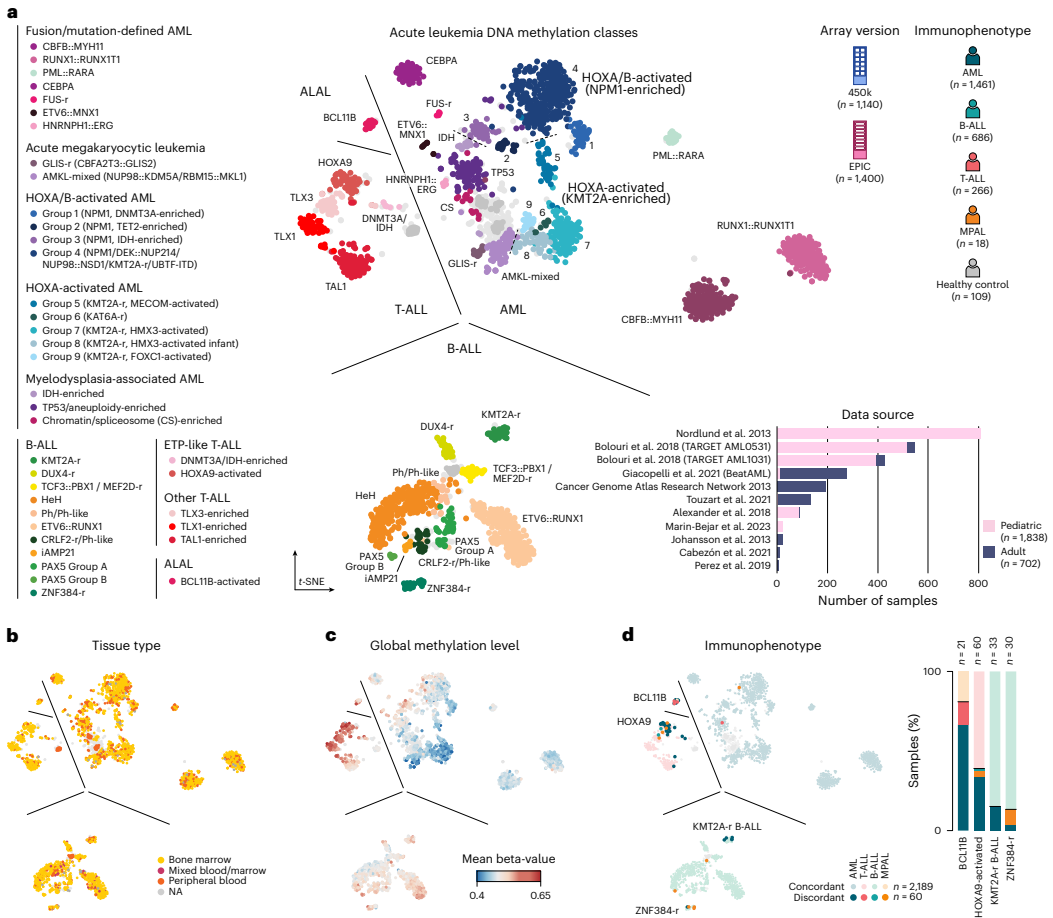
在这项发表于《Nature Genetics》的研究中,研究团队建立了急性白血病DNA甲基化参考队列,包含2,540个高质量样本,来自11个已发表的甲基化阵列数据集。通过t-SNE降维分析,研究人员观察到样本按谱系清晰分离,并在各谱系内部发现显著的表观遗传异质性。最终定义了38个甲基化类别,其中25个与既往研究一致,4个通过细分现有类别产生,9个为首次报道。值得注意的是,甲基化分类与免疫表型分型结果高度一致(97.3%吻合),但对于混合表型急性白血病(MPAL) cases,71.4%被重新分类为单一谱系,表明甲基化特征更能反映分子驱动而非表面标志物。
研究特别聚焦AML的异质性,发现了8个新甲基化类别。遗传驱动定义的类别(如PML::RARA、RUNX1::RUNX1T1等)显示出95.5%的分子改变符合率,而CEBPA突变病例中,位于CEBPA甲基化类别外的13例均伴有其他白血病相关基因突变,且缺乏典型bZIP结构域插入缺失,临床预后显著较差。这印证了转录组研究的类似发现,表明表观遗传特征能够补充遗传学分类的不足。
HOX激活的AML类别尤其引人注目。研究不仅确认了NPM1突变与KMT2A重排的关联,还发现了这些遗传改变分布于多个不同的HOX甲基化类别中。在HOXA/B激活的第4组中,24.7%病例缺乏已知遗传驱动,但95.2%的病例显示强烈的HOXA9和HOXB5表达,提示甲基化分类可能直接检测HOX激活状态,为menin抑制剂治疗提供线索,无论其遗传驱动如何。
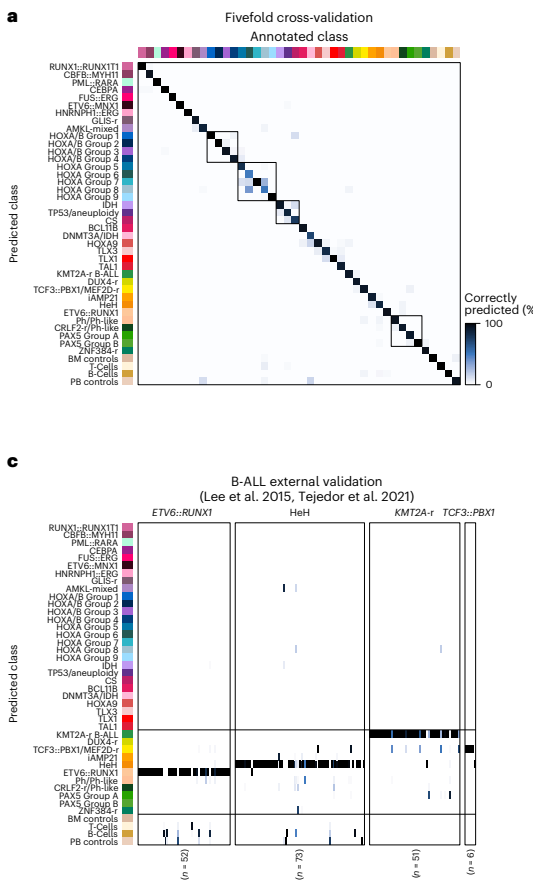
基于这一参考队列,团队开发了神经网络分类器MARLIN(甲基化与AI引导的白血病亚型快速推断系统)。该网络使用357,340个高质量甲基化位点作为输入,包含两个隐藏层,输出层对应42个类别(38个白血病类别+4个对照类别)。关键创新是在每个训练周期随机丢弃99%的输入CpG位点,使模型适应稀疏数据。交叉验证显示,MARLIN在谱系、甲基化类别和类别家族水平的F1分数分别达0.99、0.91和0.96。即使在97%稀疏度(相当于10,720个CpG位点)下,性能仍保持稳定。
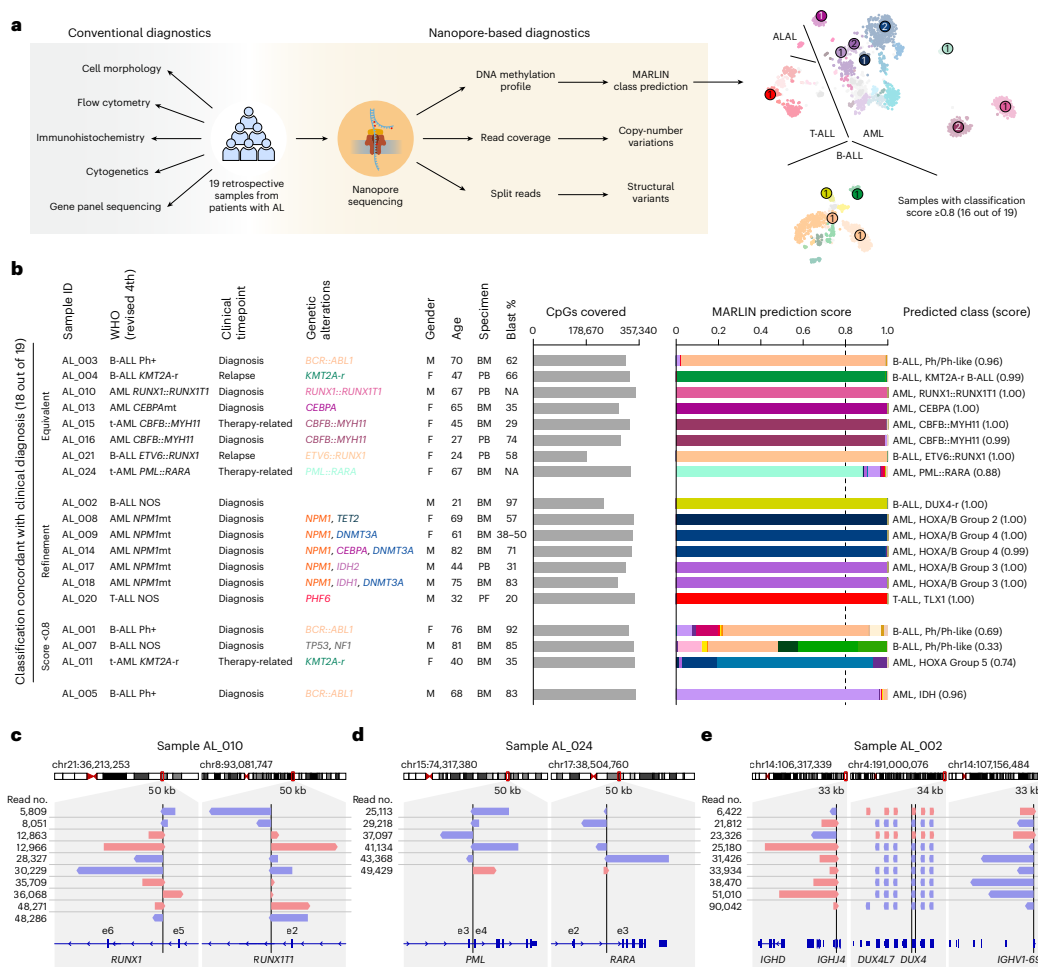
在纳米孔测序回顾性队列(19例)中,16例高置信度预测样本中的15例(93.8%)与常规病理评估一致。两个融合定义AML病例(AL_010和AL_024)通过高覆盖度PromethION测序证实了RUNX1::RUNX1T1和PML::RARA融合。一例CEBPA突变病例(AL_014)因伴有NPM1和DNMT3A共突变,被正确归类为HOXA/B激活第4组而非CEBPA类别。一例B-ALL病例(AL_002)通过甲基化分类发现了具有预后意义的隐匿性DUX4重排。唯一不一致病例(AL_005)被预测为AML IDH富集类别,而临床诊断为Ph+(p190) B-ALL,后续检查发现7q和16q染色体缺失(髓系肿瘤常见)及髓系抗原CD13/CD33共表达,提示可能为谱系模糊病例。
扩展验证中,15例多重测序样本的10例高置信度预测全部与病理结果吻合。健康供体和骨髓增生异常综合征(MDS)样本分析表明,MARLIN能区分MDS与急性白血病,但也提示了应用限制——不应将分类器用于未达到急性白血病标准诊断的病例。
研究最后验证了实时分类可行性。首例前瞻性病例(RTC_001)在样本接收后56分钟处理加40分钟测序内,即获得AML TP53/非整倍性富集类别的高置信度预测,4天后常规诊断确认AML伴TP53突变。第二例病例(RTC_002)同样在95分钟内获得HOXA/B激活第2组(NPM1/TET2富集)预测,与后续发现的NPM1、TET2和FLT3-ITD突变及单核分化特征一致。其余三例前瞻性病例也分别获得准确分类。
技术方法方面,研究主要采用:1)从公共数据库获取11个甲基化阵列数据集(共2,540样本)构建参考队列;2)使用t-SNE降维和半监督聚类定义甲基化类别;3)开发MARLIN神经网络模型,使用keras框架训练;4)回顾性和前瞻性临床样本纳米孔测序(MinION/PromethION平台);5)通过modkit工具提取甲基化呼叫并输入MARLIN预测;6)使用Panel测序和细胞遗传学分析验证结果。

研究结果部分显示,综合参考队列成功捕捉了急性白血病的全谱系多样性。甲基化分类与免疫表型高度一致,但对特定遗传驱动病例(如ZNF384重排、PICALM::MLLT10、BCL11B激活等)更反映分子本质而非表面标志物。AML分析揭示了超越遗传学分类的异质性,特别是HOX激活类别与menin抑制剂治疗响应的潜在关联。

MARLIN分类器在交叉验证中表现出色,即使在高稀疏数据下仍保持稳定性能。纳米孔测序回顾性队列验证了其临床实用性,不仅与常规诊断高度一致,还提供了更精细的分子分型。实时分析证明该方法能在2小时内提供初步分类,极大缩短诊断时间。
研究结论表明,DNA甲基化谱为急性白血病分类提供了强大工具,能够补充甚至增强标准诊断检测。通过纳米孔测序与MARLIN结合,能够在极短时间内生成可靠的分子分类,解决传统诊断的盲点,特别是对于隐匿性重排或谱系模糊病例。虽然当前参考队列可能对某些罕见亚型覆盖不足,且不包含治疗后或复发样本,但这一框架为临床急性白血病诊断提供了全新范式,未来可通过扩大样本量和纳入更多疾病实体不断完善。
这项研究的重要意义在于将表观遗传分类从概念验证推向临床实践,首次实现了急性白血病的实时表观基因组分析。其技术路线特别适合资源有限环境,有望使分子诊断服务惠及更广泛医疗机构。未来随着更多验证研究开展,甲基化分类可能成为急性白血病标准诊断的重要组成部分,为患者提供更全面、及时的治疗决策信息。
 生物通微信公众号
生物通微信公众号
生物通 版权所有